新一代平行分析的SPR生物传感器
摘要:基于表面等离子体共振的生物传感器是一种非标记的,实时生物分子相互作用检测工具。由于技术的限制,早先的SPR技术采用顺序分析的方式。现在,最新一代的SPR仪采用平行分析的方式,单次进样就可以测定1-6对相互作用的动力学。与传统技术相比,平行分析具有数据质量好、基线联动和通量高的优点。
表面等离子体共振(Surface Plasmon Resonance, SPR)技术研究生物分子以及和小分子药物之间的相互作用,具有非标记、实时检测和样品用量小的特点,因而在基础生命科学、医学研究和药物开发中应用越来越广泛[1],几乎成为研究生物分子相互作用必不可少的一种方法。
基于SPR技术的生物传感器,只有大约20年的历史。根据美国Utah大学David Myszka教授的相关文献,我们可以将SPR生物传感器的发展划分成5代,其中最先进的一代传感器具有高通量和平行分析的特点[2]。美国Bio-Rad公司最新推出的一款ProteOn XPR36蛋白相互作用阵列系统,就是其中的优秀代表,本文将着重介绍它的技术特点以及给互做研究带来的提升。
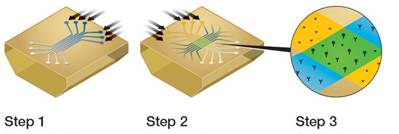
与传统的SPR生物传感器一样,最新一代技术也是将一个分子固定在芯片上,叫做ligand,另一个分子在溶液中流过ligand,叫做analyte。所不同的是两者的工作方式。以Bio-Rad公司的ProteOn XPR36系统为例,它有一套非常独特的流动池,称之为MCM(Multi- Channel Module)。MCM上有6条平行的凹槽,它先以垂直方向扣在芯片表面上,就形成了垂直方向上的6条平行通道,用以标记ligand,如下图“Step 1”。随后MCM旋转90°后再芯片表面结合,就形成6条与原先交叉的通道,可以同时流过6个不同浓度的analyte,如下图“Step 2”。这样两组通道交叉形成36个互做位点,如下图“Step 3”所示,可以实现一次analyte注入就完成它和最多6个ligand结合的动力学常数(Kon、Koff)和解离平衡常数(KD)的测定[3],而无需反复再生ligand。这种分析方式就称之为平行分析。而此前的SPR传感器芯片上不管有多少位点,都只能一次让一个浓度的analyte溶液流过芯片,叫做顺序分析。
很多人会直观地认为,平行分析极大提高了通量。这固然不错,但除了一些药厂进行药物筛选以外,对于大多数实验室而言,平行分析的最大好处是方便了实验条件的摸索,提高了数据质量。传统的SPR传感器采用顺序分析,一次注入一个浓度的analyte,在两次注入之间,需要用酸、碱、高浓度盐等试剂把和ligand结合的analyte全部洗脱下来,叫做再生。再生条件的摸索经常是一件很困难的事,过于温和的试剂不能洗脱干净,强烈一些的试剂又很容易破坏ligand,从而使最终动力学数据与实际发生偏离,有时偏离还非常明显,相差可达100倍以上。
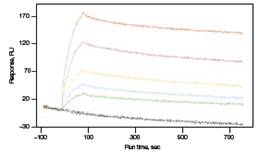
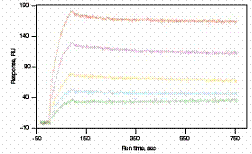
平行分析的另外一大好处是基线联动,使基线更平稳,信号更准确。对于通过捕获方式标记ligand或者存在明显非特异性反应的实验来讲,基线是不平的,而且每次再生后的基线漂移水平都不一样,很难通过一个固定的校准参数进行校准。而ProteOn系统采用的平行分析方式,可以在流过样品的同时,留出一个通道走缓冲液,观察基线漂移的情况,然后通过软件处理实时校准基线,就能得到基线平稳的、不含非特异性反应的信号。下图就是一个抗体筛选的例子。芯片上首先标记抗小鼠IgG,用来捕获培养上清中的单抗,然后流过抗原,测量单抗和抗原的结合力。因为单抗和抗原在结合的时候,单抗本身还在跟芯片上的IgG逐渐解离,使基线不断降低,而且降低的速率在不同的时间也不均匀;所以只有通过基线联动,才能知道什么时候基线在什么位置,如下左图,从而通过软件校准基线,如下右图。如果用传统的SPR传感器反复再生,就无法保证每次注入时的基线漂移完全一样,自然也就得不到准确的结果。
总之,新一代SPR生物传感器突破了传统的顺序分析模式,提高了数据质量,加快了分析速度,而且还可以降低使用成本。它把广大科研工作者从条件摸索的繁琐劳动中解放出来,从而得到了科研工作者的青睐。
[1] Rich RL and Myszka DG, Survey of the year 2004 commercial optical biosensor literature, J Mol Recognit 18, 431–478 (2005)
[2]Rich RL and Myszka DG, Higher-throughput, label-free, real-time molecular interaction analysis, Anal Biochem 361, 1–6 (2007)
[3]Tsafrir B., et al, The ProteOn XPR36TM Array System—High Throughput Kinetic Binding Analysis of Biomolecular Interactions, Cel. Mol. Bioengineering, Vol. 1, No. 4, December 2008, 216–228
